O próximo passo importante foi dado por Willem P. C. Stemmer, um pesquisador holandês e empreendedor, que morreu em 2013. Ele introduziu ainda uma outra dimensão à Evolução Dirigida de Enzimas: realiza-la em tubos de ensaio.
Cruzamento – para uma evolução mais estável
Um pré-requisito para a evolução natural é que os genes de diferentes indivíduos sejam misturados por meio de cruzamentos ou polinização, por exemplo. Propriedades benéficas podem, então, ser combinadas e dar origem a organismos mais robustos. Ao mesmo tempo, mutações em genes menos funcionais podem desaparecer de uma geração à outra.
Willem Stemmer usou o equivalente em tubo de ensaio ao cruzamento: recombinação (embaralhamento) do DNA. Em 1994, ele demonstrou que era possível cortar diferentes versões de um gene em pequenas partes e então, com auxílio dos instrumentos da tecnologia de DNA, montar um quebra-cabeças com as peças formando um gene completo, um que seria um mosaico das versões originais.
Usando vários ciclos de embaralhamento de DNA, Willem Stemmer modificou uma enzima a tal ponto que ela se tornou muito mais eficiente do que a enzima original. Isso mostrou que embaralhar genes – o que os pesquisadores chamam de recombinação genética – pode resultar em uma evolução ainda mais eficiente de enzimas.
- Mutações randômicas são introduzidas no gene da enzima que será modificada.
- Os genes são inseridos em bactérias, que os usam como receitas e produzem enzimas randomicamente mutantes
- As enzimas modificadas são testadas. Aquelas que são mais eficientes em catalisar a reação química desejada são selecionadas.
- Novas mutações randômicas são introduzidas nos genes para as enzimas selecionadas. O ciclo começa novamente.
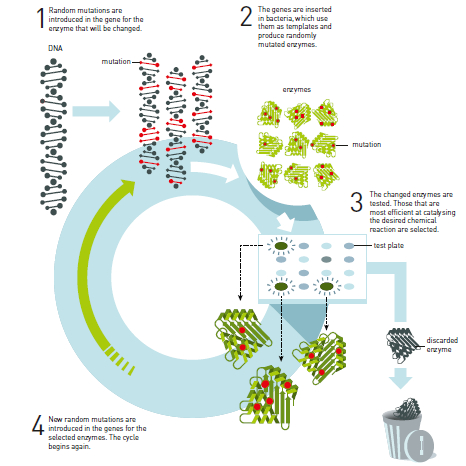
Figura 2. O princípio subjacente da evolução dirigida de enzimas. Após alguns ciclos de evolução dirigida, uma enzima pode ser milhares de vezes mais eficiente.
Novas enzimas produzem biocombustíveis sustentáveis.
Os instrumentos da Tecnologia de DNA tem sido refinados desde o início dos anos 1990s, e os métodos usados na evolução dirigida se multiplicaram. Frances Arnold tem estado nas primeiras linhas desses desenvolvimentos; as enzimas atualmente produzidas em seu laboratório podem catalizar reações químicas que nem mesmo existem na natureza, produzindo materiais inteiramente novos. Suas enzimas ajustadas sob medida também se tornaram importantes instrumentos na produção de várias substâncias, tais como as drogas farmacêuticas. As reações químicas foram aceleradas, produzem poucos subprodutos ou resíduos e, em alguns casos, foi possível excluir os metais pesados necessários na química tradicional, e assim reduzindo consideravelmente os impactos ambientais.
Algumas coisas ocorrem em ciclos: Frances Arnold está novamente trabalhando com a produção de energia renovável. Seu grupo de pesquisa desenvolveu enzimas que transformam açúcares simples em isobutanol, uma substância altamente energética que pode ser usada para a produção tanto de biocombustíveis quanto plásticos verdes. Uma meta a longo prazo é produzir combustíveis para um setor de transporte mais amigável ambientalmente. Combustíveis alternativos – produzidos pelas proteínas de Arnold – podem ser usadas em carros e aviões. Deste modo, suas enzimas estão contribuindo para um mundo mais verde.
E para avançarmos para a segunda parte da história do Prêmio Nobel de Química de 2018, onde a evolução dirigida levou a drogas farmacêuticas que podem neutralizar toxinas, combater a progressão de doenças autoimunes e, em alguns casos, até mesmo curar câncer metastático. É aqui onde um papel vital é desempenhado por um pequeno vírus que infecta bactérias e pelo método conhecido como sistema de fagos (phage display).
Smith usa Bacteriófagos
Como ocorre muitas vezes, a Ciência toma caminhos imprevisíveis. Na primeira metade dos anos 1980s, quando George Smith começou a usar bacteriófagos – vírus que infectam bactéria – foi principalmente com a esperança de que eles pudessem ser usados para clonar genes.
A tecnologia de DNA ainda era muito jovem e o genoma humana ainda era um continente não descoberto. Os pesquisadores sabiam que ele continha todos os genes necessários para produzir as proteínas do corpo, mas identificar o gene específico de uma proteína particular era mais difícil do que achar uma agulha no palheiro. Entretanto, haveria enormes benefícios para o cientista que descobrisse isso. Usando os novos instrumentos da genética da época, o gene poderia ser inserido em bactérias que – com um pouco de sorte – pudesse produzir em massa a proteína a ser estudada.
O processo completo era chamado de clonagem de gene e a ideia de George Smith foi de que os pesquisadores pesquisando genes poderiam usar bacteriófagos de um modo engenhoso e inovador.
Bacteriófagos – um elo entre uma proteína e seu gene desconhecido
Bacteriófagos são simples por natureza. Eles consistem de uma pequena porção de material genético encapsulado por proteínas protetoras. Quando eles são duplicados, eles injetam seu material genético em bactérias e sequestram (tomam emprestado) seu metabolismo. A bactéria, então, produz novas cópias do material genético do fago e as proteínas que formam a cápsula, que formam novos vírus.
A ideia de George Smith era de que os pesquisadores pudessem ser capazes de usar a construção simples dos fagos para encontrar o gene desconhecido de uma proteína. Nessa época, grandes bibliotecas moleculares estavam disponíveis que continham massas de fragmentos de vários genes desconhecidos. Sua ideia era que estes fragmentos de genes desconhecidos podiam ser reunidos com o gene de uma das proteínas na cápsula do fago. Quando novos fagos fossem produzidos, as proteínas do gene desconhecido terminariam sobre a superfície do fago como parte da proteína da cápsula (figura 3).
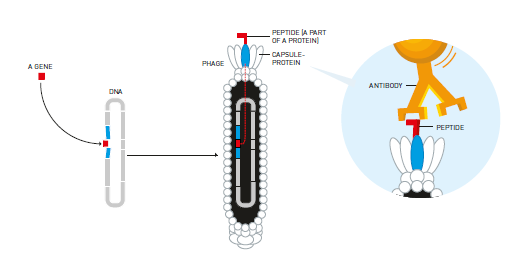
- Smith introduziu um gene no gene de uma proteína da cápsula do fago. O fago DNA foi então inserido na bactéria que produziu novos fagos.
- O peptídeo produzido com o gene introduzido termina como parte da proteína da cápsula sobre a superfície do fago.
- Smith foi capaz de pescar o fago usando um anticorpo projetado para capturar o peptídeo.
- Como um bônus, ele obteve o gene do peptídeo.
Figura 3. Sistema Fago (Phage display): George Smith desenvolveu seu método para encontrar genes desconhecidos de uma proteína conhecida.
Anticorpos podem “pescar” a proteína certa
Isto vai resultar em uma mistura de fagos que carregam múltiplas proteínas diferentes sobre sua superfície.
No estágio seguinte – George Smith postulou – os pesquisadores seriam capazes de usar anticorpos para pescar fagos carregando várias proteínas conhecidas desta sopa. Anticorpos são proteínas que funcionam como mísseis com alvos; eles podem identificar e se conectar a uma única proteína entre dezenas de milhares de outras com extrema precisão. Se os pesquisadores capturassem algo na sopa de bacteriófagos usando um anticorpo que eles sabiam se acoplar a uma proteína conhecida, como um produto extra, eles obteriam o até então gene desconhecido para a proteína.
Esta era uma ideia elegante e, em 1985, George Smith demonstrou que ela podia funcionar. Ele produziu um bacteriófago que carregava parte de uma proteína, um peptídeo, sobre sua superfície. Usando um anticorpo, ele foi capaz de pescar o bacteriófago que ele tinha construído de uma sopa de muitos bacteriófagos.
Por meio deste experimento, George Smith estabeleceu as fundações do que passou a ser conhecido como Sistema Fago (phage display). O método é brilhante em sua simplicidade. Sua força se baseia no fato do bacteriófago funcionar como um elo entre uma proteína e o seu gene. Entretanto, não foi dentro da clonagem de genes que o método teve o seu maior impacto; além disso, por volta de 1990, vários grupos de pesquisa começaram a usar o phage display para desenvolver novas biomoléculas. Uma das pessoas que adotaram a técnica foi Gregory (Greg) Winter e foi graças a sua pesquisa que o phage display está promovendo grandes benefícios à humanidade. Mas, para compreender porque, nós precisamos dar uma olhada mais de perto nos anticorpo.
Anticorpos podem bloquear processos de doenças
O sistema linfático humano possui células que podem produzir centenas de milhares de diferentes tipos de anticorpos. Usando um sistema bem desenvolvido, todas essas células tem sido testadas de modo que nenhum anticorpo se acople a qualquer dos vários tipos de moléculas do corpo. Entretanto, esta enorme variação assegura que haja sempre um anticorpo que se acopla a vírus ou bactérias que nos infectam. Quando um anticorpo se acopla aos mesmos, ele envia um sinal para as agressivas células imunes para destruírem os invasores.
Como os anticorpos são altamente seletivos e podem se acoplar a uma única molécula entre dezenas de milhares de outras, os pesquisadores esperam há muito que seja possível projetar anticorpos que bloqueiem vários processos de doenças no corpo e que funcionem como medicamentos. Inicialmente, para obter estes anticorpos terapêuticos, camundongos foram injetados com diferentes alvos para medicamentos, tais como proteínas de células cancerígenas. Entretanto, nos anos 1980s ficou cada vez mais claro que este método tinha limitações; algumas substâncias eram tóxicas para os camundongos e outras não resultavam em nenhuma produção de anticorpos.
Além disso, foi descoberto que os anticorpos obtidos eram reconhecidos como estranhos pelo sistema imune dos pacientes, que os atacava. Isto levava à destruição de anticorpos nos camundongos e havia o risco de efeitos colaterais para os pacientes.
Foi este obstáculo que fez Greg Winter começar a investigar o potencial do Sistema Fago de George Smith. Ele queria evitar usar camundongos e ser capaz de basear os medicamentos em anticorpos humanos porque eles seriam tolerados pelo nosso sistema imune.
Winter coloca anticorpos sobre a superfície de bacteriófagos
Anticorpos são moléculas em forma de Y; é a extremidade de cada braço que se acopla a substâncias externas.
Greg Winter reuniu a informação genética desta parte de um anticorpo do gene das proteínas da cápsula de um bacteriófago e, em 1990, ele conseguiu demonstrar que isto fazia o sítio de acoplamento do anticorpo terminar sobre a superfície da cápsula do bacteriófago. O anticorpo que ele usou era projetado para se acoplar a uma pequena molécula conhecida como phOx. Quando Greg Winter usou a phOx como um tipo de anzol de pesca molecular, ele obteve sucesso em extrair o bacteriófago com o anticorpo em sua superfície da sopa de quatro milhões de outros bacteriófagos.
Em seguida, Greg Winter mostrou que ele podia usar o Sistema Fago (phage display) na Evolução Dirigida de Anticorpos.
Ele construiu uma biblioteca de bacteriófagos com bilhões de variedades de anticorpos em suas superfícies. Desta biblioteca, ele pescou anticorpos que se acoplavam a diferentes proteínas alvos. Ele, então, randomicamente modificou sua primeira geração de anticorpos e criou uma nova biblioteca, na qual ele encontrou anticorpos com acoplamento ainda mais intenso às proteínas alvo. Por exemplo, em 1994 ele usou este método para desenvolver anticorpos que se acoplavam a células cancerígenas com um alto nível de especificidade.
O primeiro medicamento mundial baseado em um anticorpo humano
Greg Winter e seus colegas fundaram uma empresa baseada no Sistema Fago (phage display) de anticorpos. Nos anos 1990s, a empresa desenvolveu um medicamento inteiramente baseado em um anticorpo humano: o adalimumab. O anticorpo neutraliza uma proteína, a TNF-alpha, que comanda a inflamação em muitas doenças autoimunes. Em 2002, o medicamento foi aprovado para o tratamento de artrite reumatoide e é usada atualmente também para o tratamento de diferentes tipos de psorises e doenças inflamatórias intestinais.
O sucesso da adalimumab instigou um desenvolvimento significativo na indústria farmacêutica e
o Phage Display tem sido usado para produzir anticorpos cancerígenos, entre outros. Um deles libera as células assassinas do corpo de modo que elas possam atacar células tumorais. O crescimento dos tumores desacelera e, em alguns casos, até mesmo pacientes com metástase de câncer foram curados, o que é um grande avanço histórico na cura do câncer.
Outro anticorpo farmacêutico que foi aprovado neutraliza a toxina bacteriana que causa o anthrax, enquanto outro reduz a doença autoimune conhecida como lupus; muito mais anticorpos estão atualmente passando por testes clínicos, por exemplo para combater a doença de Alzheimer.
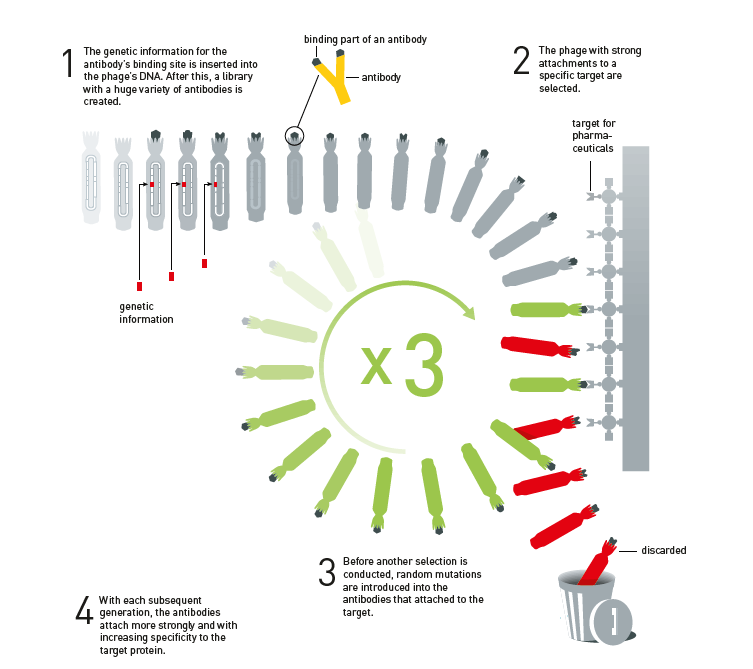
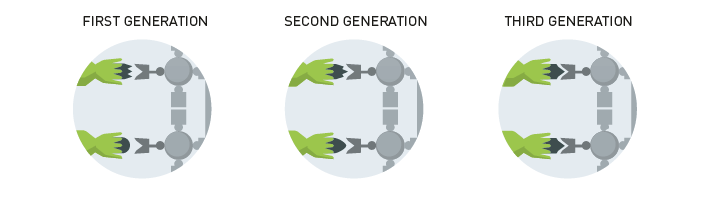
Figura 4. O princípio da Evolução Dirigida de Anticorpos usando Sistema Fago (phage display). Este método é usado para produzir novos medicamentos.
Início de uma nova era na Química
Os métodos que os Laureados do Prêmio Nobel de Química de 2018 desenvolveram são, atualmente, sendo internacionalmente desenvolvidos para promover uma Indústria Química Mais Verde, produzir novos materiais, manufaturar biocombustíveis sustentáveis, mitigar doenças e salvar vidas. A Evolução Dirigida de Enzimas e o Sistema Fago de anticorpos possibilitaram que Frances Arnold, George Smith e Greg Winter abrissem as portas para enormes benefícios à humanidade e estabelecer as fundações para uma revolução na Química.
